- Labor
- Labormedizin
- Analysesoftware
- SoftGenetics, LLC.
Software zur NGS-Sequenzierung NextGENe®AnalyseLabor
Zu meinen Favoriten hinzufügen
Zum Produktvergleich hinzufügen
Eigenschaften
- Funktion
- Analyse
- Anwendung
- zur NGS-Sequenzierung, Labor
Beschreibung
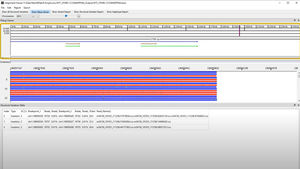
Die NextGENe-Software ist der perfekte analytische Partner für die Analyse von Desktop-Sequenzierungsdaten, die von Illumina® iSeq-, Miniseq-, MiSeq-, NextSeq-, HiSeq- und NovaSeq-Systemen, Ion Torrent Ion GeneStudio S5-, PGM- und Proton-Systemen sowie anderen Plattformen erzeugt werden.
Die NextGENe-Software läuft auf einem Windows®-Betriebssystem, das eine biologenfreundliche "Point & Click"-Benutzeroberfläche bietet. Sie erfordert keine Skripterstellung oder andere bioinformatische Unterstützung, die bei der Verwendung von Programmen wie CLC Genomics Workbench, Lasergene's SeqMan Pro sowie akademischer Software wie MAQ & SOAP, Top Hat, BWA & Bowtie oft erforderlich sind.
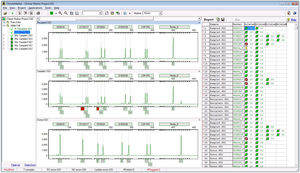
Die NextGENe-Software nutzt einzigartige plattformspezifische Technologien in einem eigenständigen Multiapplikationspaket. Die NextGENe-Software enthält Analysemodule für:
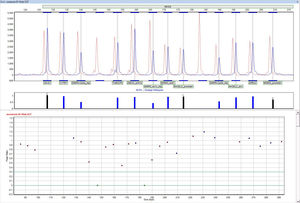
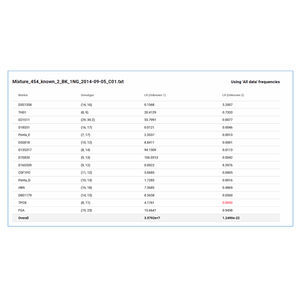
SNP/Indel-Erkennung
Keimbahn
Somatische
Exom-Erfassung, Whole Exome Sequencing (WES)
Vollständige Genomsequenzierung (WGS)
Strukturelle Varianzanalyse (einschließlich Nachweis von Fusionsgenen)
Familienbasierte und Trio-Vergleiche
Tumor-Normal-Vergleiche
Nachweis von Kopienzahlvariationen (CNV) und Alternative zum MLPA-ähnlichen Screening
de novo-Zusammensetzung
Transkriptom; Analyse des alternativen Spleißens und der Transkript-Expressionsniveaus
ChIP-Seq, digitale Genexpression (DGE), miRNA-Analyse & Quantifizierung & Metagenomik
Die NextGENe-Software wurde in einer biologenfreundlichen Windows®-Umgebung entwickelt, die den Bedarf an zusätzlichen Bioinformatik-Ressourcen und -Kosten erheblich reduziert. NextGENe Software nutzt kostengünstige 64-bit Windows OS basierte Hardware. (Klicken Sie hier für die vorgeschlagene Hardwarekonfiguration)
---
Verwandte Suchbegriffe
- Analysesoftware
- Viewer-Software
- Steuerungssoftware
- Laborsoftware
- Automatisierte Software
- Monitoring-Software
- Projektentwicklungssoftware
- Überwachte Software
- Import-Software
- Server-Software
- Software für Forschungszwecke
- Interpretations-Software
- Molekularbiologie-Software
- Genetik-Software
- Software zur NGS-Sequenzierung
- NGS-Software
- Software für klinische Forschung
- Software für Genomforschung
- Software zur Sequenzierung
- Software / Kapillar Elektrophorese
* Die Preise verstehen sich ohne MwSt., Versandkosten und Zollgebühren. Eventuelle Zusatzkosten für Installation oder Inbetriebnahme sind nicht enthalten. Es handelt sich um unverbindliche Preisangaben, die je nach Land, Kurs der Rohstoffe und Wechselkurs schwanken können.