Analysesoftware GeneMarker®HTS SteuerungReportingNGS

Zu meinen Favoriten hinzufügen
Zum Produktvergleich hinzufügen
Eigenschaften
- Funktion
- Analyse, Steuerung, Reporting, NGS
- Anwendung
- medizinisch, für DNA-Sequenzierung
Beschreibung
Die GeneMarker®HTS-Software bietet einen validierten, rationalisierten Arbeitsablauf für forensische mitochondriale, STR- und Y-STR-Fälle sowie für die medizinische Forschung an mitochondrialer DNA von massiv-parallelen Squencing-Plattformen (MPS) - wie Illumina® und Ion Torrent® - in einem einfach zu bedienenden Windows®-Betriebssystem.
GeneMarkerHTS (High Through-put Sequence) Forensic Analysis Software für Daten von Next Generation Sequence (NGS), Massively Parallel Sequencing (MPS) Plattformen, einschließlich Illumina® und Ion Torrent®
Mitochondriale DNA-Analyse
Analyse des gesamten Genoms, von HVI/HVII und Kontrollregionen
Einzigartige Alignment-Technologie ∙ Motiv-Konsensus
Forensische Nomenklatur
Einfacher Upload zu EMPOP
STR-Analyse
Autosomal & Y-STR
Forensische Nomenklatur
Genotyp- & SNP-Berichterstattung
Konkordanz
Validierte, leicht zu bedienende Windows®-Schnittstelle
Kompatibel mit den wichtigsten Chemielabors und Plattformen
Audit Trail & Benutzerkontrolle
Umfassende Berichtsoptionen
Die GeneMarkerHTS-Software umfasst:
Audit-Trail-Fähigkeit
Benutzerverwaltung
Individuell anpassbare Anzeige und Berichterstattung zum Schutz persönlicher Gesundheitsinformationen (PHI)
Profilvergleichsfunktionen
Hochladen zu EMPOP
Schnelle Analyse - Ergebnisse in wenigen Minuten
mtDNA:
30 MiSeq-Dateien mit ganzen mtDNA-Chromosomen mit einer durchschnittlichen Abdeckungstiefe von 10.000 wurden in 90 Minuten (3 Minuten pro Probe) aligniert.
200 ganze mtDNA-Chromosomendateien mit einer durchschnittlichen Abdeckungstiefe von 10.000 wurden in 16 Stunden aligniert.
STR/Y-STR
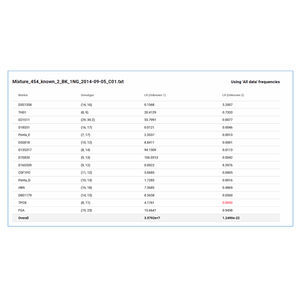
Die Ergebnisse der GeneMarker HTS-Software stimmten zu 99,74 % mit den CE-Allel-Calls von 20.000 beprobten GeneMarker-Loci überein.
Allel-Calls und Iso-Allele der Proben
---
VIDEO
Kataloge
Für dieses Produkt ist kein Katalog verfügbar.
Alle Kataloge von SoftGenetics, LLC. anzeigenVerwandte Suchbegriffe
* Die Preise verstehen sich ohne MwSt., Versandkosten und Zollgebühren. Eventuelle Zusatzkosten für Installation oder Inbetriebnahme sind nicht enthalten. Es handelt sich um unverbindliche Preisangaben, die je nach Land, Kurs der Rohstoffe und Wechselkurs schwanken können.