- Labor
- Labormedizin
- Datenverwaltungssoftware
- PhenoSystems SA

- Produkte
- Kataloge
- News & Trends
- Messen
Software zur NGS-Sequenzierung GensearchNGSAnalyseDatenverwaltungDiagnose
Zu meinen Favoriten hinzufügen
Zum Produktvergleich hinzufügen
Eigenschaften
- Funktion
- Analyse, Datenverwaltung, Diagnose
- Anwendung
- zur NGS-Sequenzierung, Labor
Beschreibung
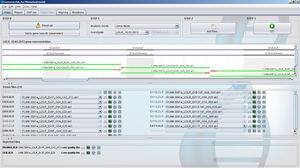
GensearchNGS ist eine integrierte Softwarelösung für die Analyse von DNA-Seq-Daten von häufig verwendeten NGS-Geräten wie Roche/454, Illumina, Ion Torrent und Proton. Es wurde in enger Zusammenarbeit mit führenden akkreditierten Diagnostikgruppen in Europa und mit Unterstützung des NMDchip FP7-Projekts konzipiert und entwickelt.
Es integriert alle Schritte vom Import und der Filterung von Lesesequenzen, der Barcodeaufteilung, der Ausrichtung, der Variantenerkennung und der Annotation bis hin zum Endbericht der erkannten Varianten. Varianten können mit verschiedenen öffentlichen und privaten Datenquellen (Ensembl, Alamut, Genome Trax,...) annotiert werden. Sie können durch logische Kombinationen zwischen den Proben für Familienstudien gefiltert werden.
Hauptmerkmale:
Benutzerfreundliche Software zur Resequenzierung von NGS-Projekten in einer diagnostischen Umgebung, die auf Windows-, Linux- und MacOS-Systemen läuft.
Verbindet*) mit Varianten-Interpretationswerkzeugen und Datenbanken wie z.B: Ensembl, dbSNP, Alamut, Clinvar, Wiki-Pfade, Gen-Ontologie*).
Filtern Sie Varianten über Proben hinweg (z.B. Varianten, die mehreren Proben gemeinsam sind oder Varianten einer Probe von einer anderen Probe ausschließen).
Patientenzentriertes Datenbanksystem zur Verwaltung von Sequenzdaten und validierten Varianten.
Individuelle Anpassung möglich, um Daten in Ihr Laborverwaltungssystem zu exportieren.
Möglichkeit, Ihre Varianten auf Clinvar*) zu veröffentlichen.
Gemeinsam mit akkreditierten Laboratorien entwickelt.
Für größere Projekte (WES/WGS): Möglichkeit, mehrere PCs parallel zu verwenden.
Plugin-Technologie: Abgleich mit unseren proprietären oder öffentlichen Algorithmen wie Bowtie, BWA oder Stampy.
Annotation und Filterung mit HPO-, OMIM- und Orphanet-Deskriptoren und -Codes, hilfreich bei der Suche nach Mutationen in ganzen Exomen oder ganzen Genomen.
---
* Die Preise verstehen sich ohne MwSt., Versandkosten und Zollgebühren. Eventuelle Zusatzkosten für Installation oder Inbetriebnahme sind nicht enthalten. Es handelt sich um unverbindliche Preisangaben, die je nach Land, Kurs der Rohstoffe und Wechselkurs schwanken können.