- Labor
- Molekularbiologie
- Software-Modul / Analyse
- JSI medical systems
Software-Modul / zur NGS-Sequenzierung SEQNEXTAnalyseErfassungKartografie
Zu meinen Favoriten hinzufügen
Zum Produktvergleich hinzufügen
Eigenschaften
- Funktion
- Analyse, Erfassung, Kartografie
- Anwendung
- Labor, zur NGS-Sequenzierung
Beschreibung
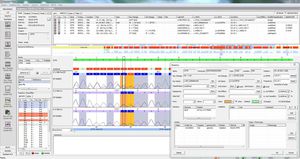
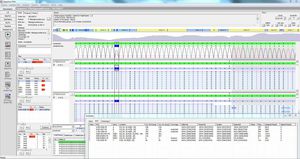
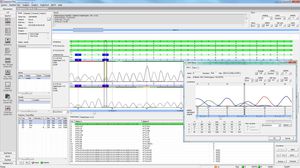
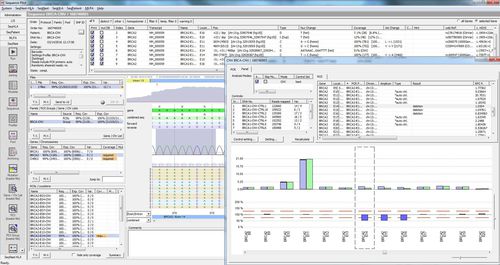
SEQNEXT ist eine leistungsstarke und benutzerfreundliche Anwendung für Mapping, Alignment und Variantenerkennung von Next Generation Sequencing-Daten. SEQNEXT kann Daten von allen gängigen Sequenzierplattformen und Kits (PCR-basiert, Anreicherung, Sequenzerfassung) analysieren. Die Visualisierung aller entdeckten Varianten wie Deletionen, Insertionen und Indels beliebiger Länge, SNPs, Repeat-Regionen sowie CNVs und Genfusionen ist übersichtlich und intuitiv.
SEQNEXT bietet Zugang zu öffentlichen SNP-Datenbanken wie dbSNP, 1000 Genomes, ClinVar, ExAC und gnomAD zur Klassifizierung und Filterung. Alle Ergebnisdaten können mit unserer Variantendatenbank für Shared Experience And Knowledge - varSEAK ausgetauscht, in laborinterne LIM-Systeme übertragen und/oder als personalisierte Patientenberichte ausgegeben werden.
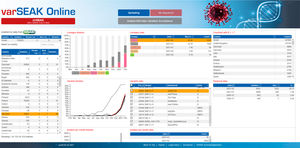
NEU Verbesserte Analyse von Virussequenzierungsdaten, z.B. SARS-CoV-2
Trio-Analyse und verbesserte Pool-Analyse
Sequence Ontology Annotationen in der Variantentabelle
Sprungoption zu varSEAK und allen verfügbaren externen DBs wie dbSNP, ClinVar, UK10K, ESP, gnomAD und ExAC
Whole Exome Sequencing (WES) Analyse
Kompatibel mit Daten von allen gängigen Next Generation Sequencing Plattformen und Kits
Einfaches Einrichten individueller Zielregionen (ROIs) durch Import von bed- oder manifest-Dateien auf Basis von hg19 und / oder hg38
Import von Genlisten zur Filterung und Analyse von WES-Daten
Analyse von fastq-, fastq.gz- oder bam-Dateien
Effiziente Standards für Mapping, Alignment, Qualität und Variantenaufruf (verwendete Algorithmen: BWA und Smith-Waterman, angepasst von JSI)
Personalisierte Einstellungen für Analyse und Variantenaufruf (≥ 0,1 %)
Hohe Sensitivität und Spezifität für die Erkennung von Deletionen, Insertionen und Indels beliebiger Länge, SNPs, Repeat-Regionen sowie CNVs und Genfusionen
---
Kataloge
SEQUENCE PILOT
2 Seiten
* Die Preise verstehen sich ohne MwSt., Versandkosten und Zollgebühren. Eventuelle Zusatzkosten für Installation oder Inbetriebnahme sind nicht enthalten. Es handelt sich um unverbindliche Preisangaben, die je nach Land, Kurs der Rohstoffe und Wechselkurs schwanken können.