- Labor
- Molekularbiologie
- Molekularbiologie-Software
- JSI medical systems
Molekularbiologie-Software virSEAKReportingImportNGS
Zu meinen Favoriten hinzufügen
Zum Produktvergleich hinzufügen
Eigenschaften
- Anwendung
- Molekularbiologie
- Funktion
- Reporting, Import, NGS
- Typ
- automatisiert
Beschreibung
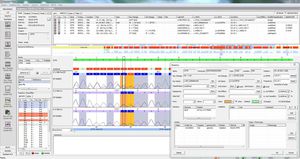
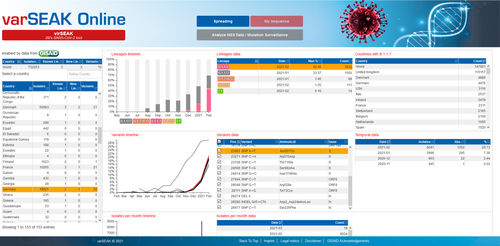
virSEAK (RUO - research use only) bietet einen schnellen und bequemen Vergleich Ihrer SARS-CoV-2-Sequenz mit den verfügbaren Sequenzen. Nach einer personalisierten Einrichtung mit Ihrem Kit laden Sie einfach Fastq- oder Fasta-Daten und erhalten eine alignierte Sequenz mit Varianten.
Zusätzlich wird Ihre Sequenz einem Schuppentierstamm (z.B. B.1.1.7) und einer GISAID-Klade (z.B. L) zugeordnet.
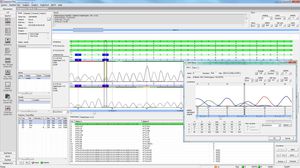
virSEAK prüft auch automatisch, ob Ihre Sequenz die Spike-Protein-Varianten N501Y und oder E484K enthält.
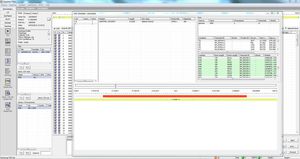
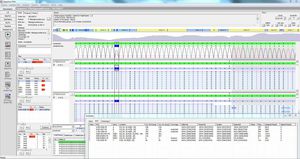
Die Werte für Abdeckung und Qualität werden angezeigt, einschließlich: erforderliche, absolute, durchschnittliche und mittlere Abdeckung, % der "Wildtyp"- und "N"-Basen. Ein Qualitätsindikator zeigt an, ob die Qualität den festgelegten Qualitätsschwellenwerten entspricht.
Außerdem ist das Tool für die NGS-Sequenzierung mit hohem Durchsatz optimiert, mit automatischen Import- und Exportfunktionen und anpassbarem Sequenztransfer. Sie können problemlos Multi-Fasta-Dateien erstellen, um die Meldung an eine zentrale Meldestelle zur Überwachung der epidemiologischen Situation und der Verbreitung von Mutationen zu verbessern (z. B. für Deutschland das Robert Koch-Institut).
virSEAK - installierbare Version:
einfacher Import von rohen fastq oder vorverarbeiteten fasta Sequenzierdateien
hochdurchsatz-NGS-Sequenzierung mit automatischem Batch-Import und -Export
automatische Warnung, wenn die Mutation(en) N501Y und/oder E484K enthalten sind
zuordnung zu einem Schuppentierstamm und einer GISAID-Klade
überprüfung auf andere Varianten mit Häufigkeiten
vergleich von Subtypen, Anzahl und Lage der Befunde, ...
sequenz und Varianten exportieren (mit Metainformationen, im Fasta- und/oder Csv-Format)
ergebnisse werden gespeichert
vollautomatische Hochdurchsatz-Sequenzierung mit anpassbarem Sequenztransfer
---
Kataloge
Für dieses Produkt ist kein Katalog verfügbar.
Alle Kataloge von JSI medical systems anzeigen* Die Preise verstehen sich ohne MwSt., Versandkosten und Zollgebühren. Eventuelle Zusatzkosten für Installation oder Inbetriebnahme sind nicht enthalten. Es handelt sich um unverbindliche Preisangaben, die je nach Land, Kurs der Rohstoffe und Wechselkurs schwanken können.